- 脂肪酸関連別記事:人類を救う理想のアブラ!? オメガ3との付き合い方(NHK健康チャンネル,2020/01/08) ※“オメガ3脂肪酸(DHA・EPA)”のDHA・EPAを以下に。
- 脂肪酸 - Wikipedia | ω-3脂肪酸 | ω-6脂肪酸
- 有機化合物の名前・基礎の基礎(本サイト)
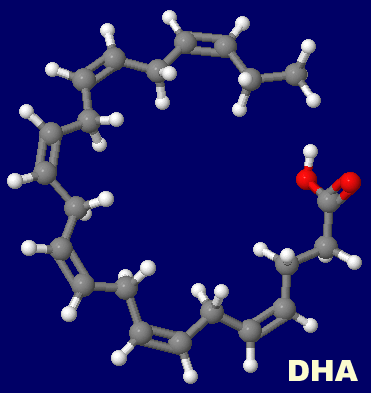
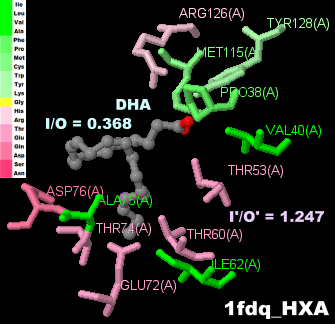
[左・中]DHA(ドコサヘキサエン酸,docosahexaenoic acid)とDHAが結合した脳の脂肪酸結合タンパク質1fdqPDBsumデータ
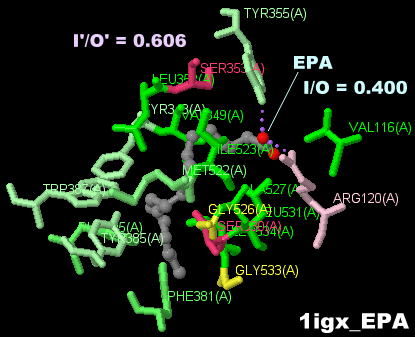
[右]EPAが結合したプロスタグランジンエンドペルオキシド合成酵素1igxのPDBsumデータ
-
●アミノ色表示の凡例
ASP GLU CYS MET LYS ARG SER THR PHE TYR ASN GLN GLY LEU VAL ILE ALA TRP HIS PRO
●酸性・中性〈芳香族〉・塩基性アミノ酸区別表示の凡例
ASP GLU GLY ALA VAL LEU ILE CYS SER THR ASN GLN PRO MET PHE TYR TRP LYS ARG HIS
●極性〈酸性・塩基性〉・非極性(疎水性)アミノ酸区別
SER THR TYR CYS ASN GLN ASP GLU LYS ARG HIS GLY ALA VAL LEU ILE PHE PRO MET TRP
●疎水性インデックス順
ARG LYS ASN ASP GLN GLU HIS PRO TYR TRP SER THR GLY ALA MET CYS PHE LEU VAL ILE
●有機概念図I/O値順(特性基 R)
ASN SER ASP GLN GLU THR ARG HIS GLY LYS TYR TRP CYS MET PRO PHE ALA VAL LEU ILE
●等電点順
ASP GLU CYS ASN PHE GLN TYR SER MET TRP VAL GLY LEU ALA ILE THR PRO HIS LYS ARG
アミノ酸および特性基の親水性・疎水性 | Log Pをポケットに!
ニューロテンシン2lneのアミノ酸配列(13塩基,有機概念図I/O値順着色)
[UV degradation - Wikipedia]
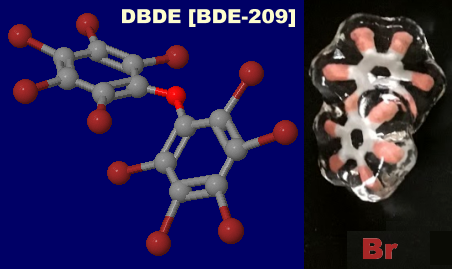
難燃剤 DBDE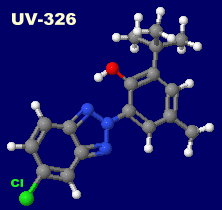
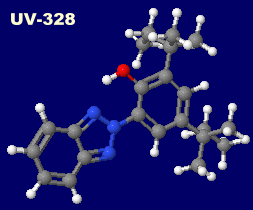
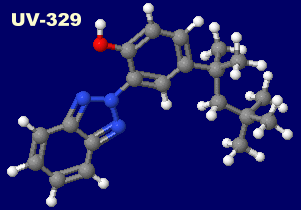
左から 紫外線吸収剤 UV-326・UV-328・UV-329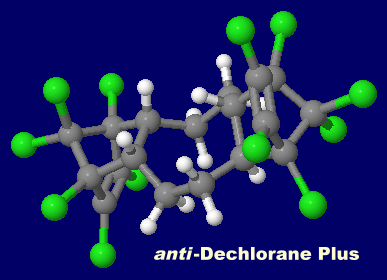
難燃剤 デクロランプラス(anti-Dechlorane Plus;他にsyn体あり)
[M2 proton channel - Wikipedia]
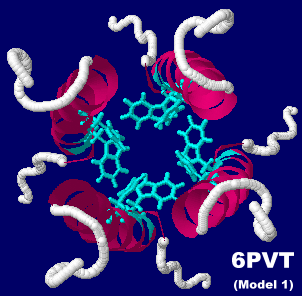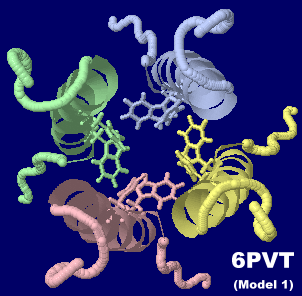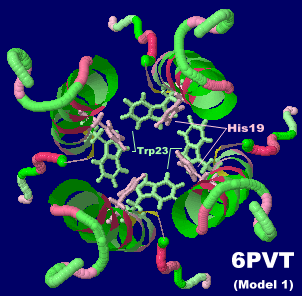
B型インフルエンザウイルスのBM2タンパク質プロトンチャネル6pvtのModel 1
[Neurotensin - Wikipedia]
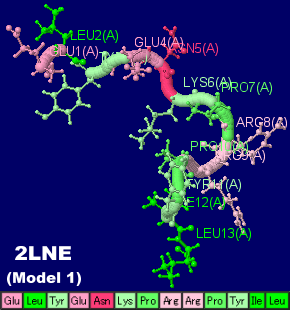
[左]ヒトのニューロテンシン2lneのModel 1
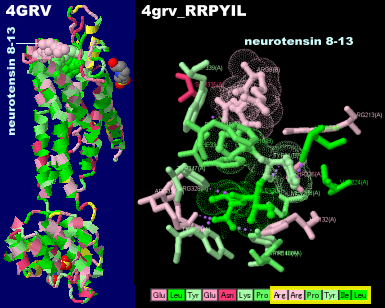
[中・右]参考(ニューロテンシン結合タンパク質例):ニューロテンシン受容体1(NTSR1,ニューロテンシン〈部分,neurotensin 8-13結合)とPDBsumデータ
Glu
Leu
Tyr
Glu
Asn
Lys
Pro
Arg
Arg
Pro
Tyr
Ile
Leu
Lithocholic Acid Amides as Potent Vitamin D Receptor Agonists(Biomolecules,2022/01/24) ※2022/03/09公開7vqp
Structure-activity relationship and crystallographic analyses of non-secosteroidal vitamin D receptor ligands bearing diphenylsilane core as a hydrophobic pharmacophore(Bioorganic & Medicinal Chemistry,2025/05/30) [NEW!] ※2025/06/11公開9m10・9m11・9m13・9m14(下掲)・9m15・9m16・9m17・9m18・9m19・9m1a・9m1b・9m1c・9m1d
[Fluorophore - Wikipedia,Vitamin D - Wikipedia,Calcitriol receptor - Wikipedia]
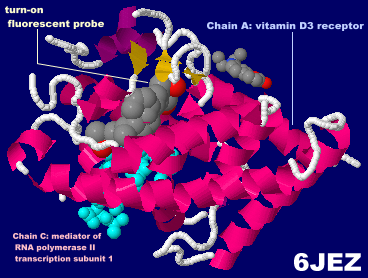
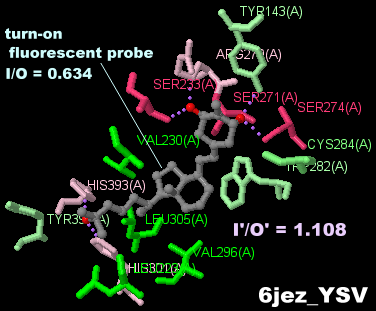
ターンオフ蛍光プローブが結合したビタミンD3受容体6jezとPDBsumデータ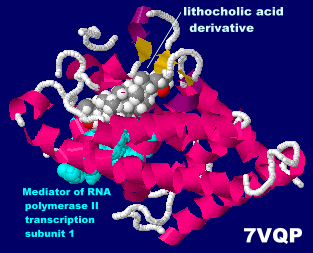
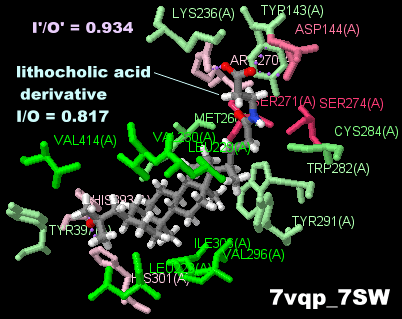
リトコール酸誘導体が結合したビタミンD3受容体7vqpとPDBsumデータ(リガンドにH原子付加)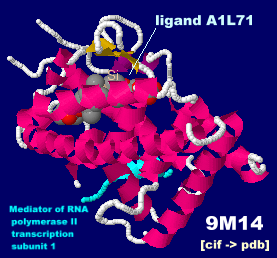
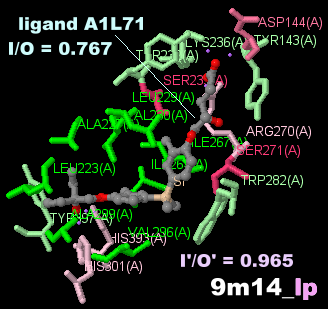
ジエチル-ジ-m-トリルシラン誘導体が結合したビタミンD3受容体9m14とPDBsumデータ
[Hemoglobin - Wikipedia]
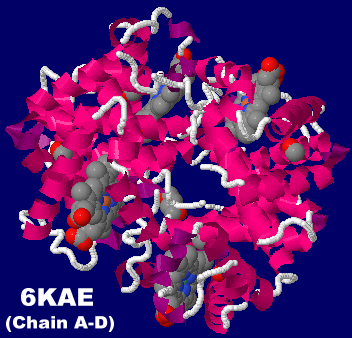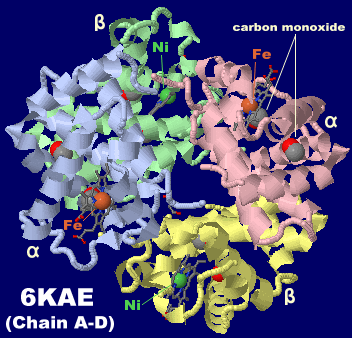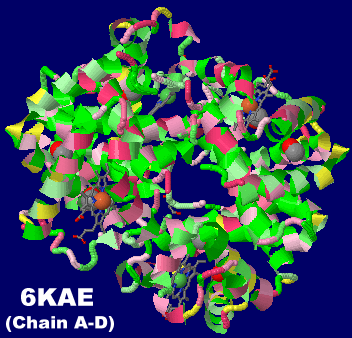
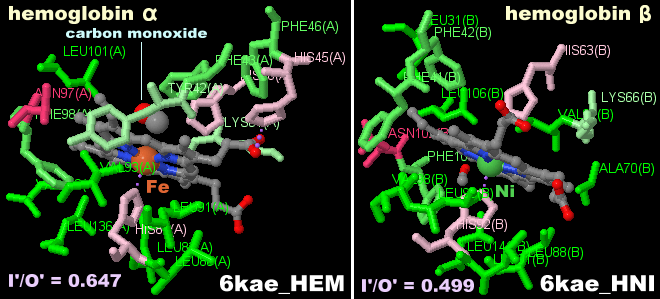
一酸化炭素が結合した Ni(II)-Fe(II)混成ヘモグロビン6kaeのChain A-DとPDBsumデータ(Chain A〈CO結合〉・B)
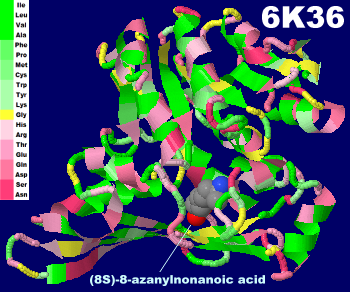
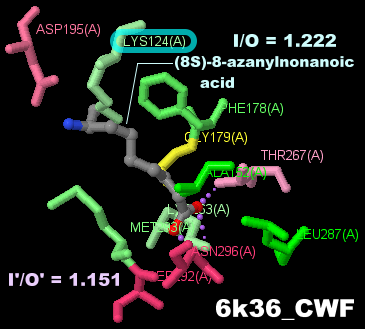
新規生合成酵素BioU 6k36とPDBsumデータ(Lys124については上掲論文・プレスリリース参照)
[Sodium channel - Wikipedia,Tetrodotoxin - Wikipedia]
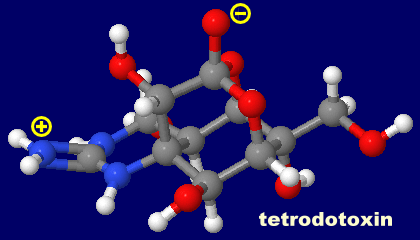
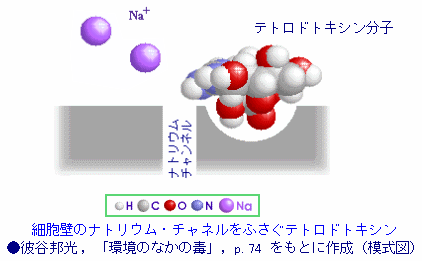
フグ毒テトロドトキシン(tetrodotoxin)とその作用機序模式図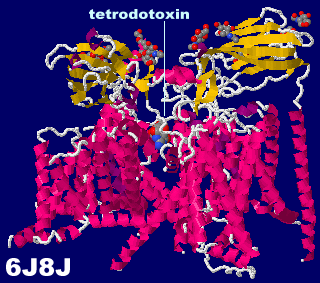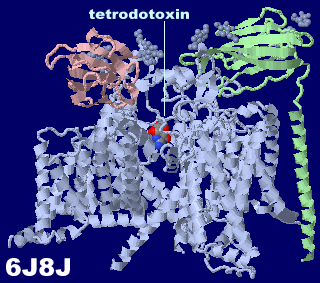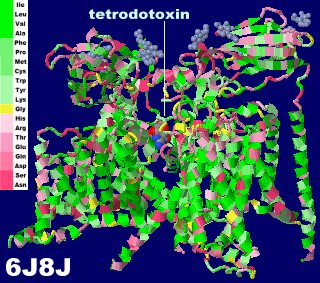
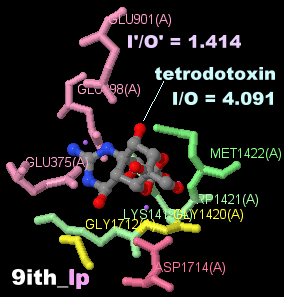

[左]テトロドトキシンが結合した電位依存性ナトリウムチャネルNav1.7 6j8jとPDBsumデータ
[右]同Nav1.5 9ithとPDBsumデータ
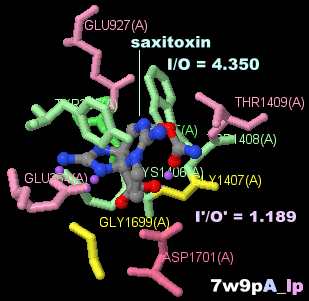
[左]サキシトキシン(saxitoxin)が結合した電位依存性ナトリウムチャネルNav1.7 7w9pのChain AとPDBsumデータ
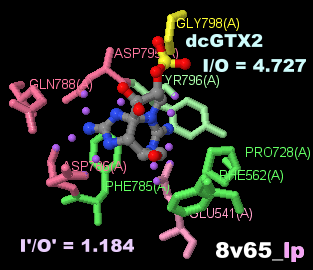
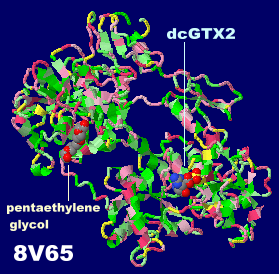
[右]参考データ:サキシトキシン同族体のdcGTX2が結合したサキシフィリン8v65とPDBsumデータ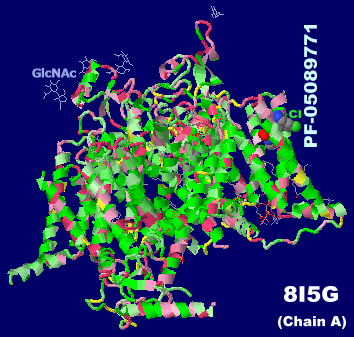
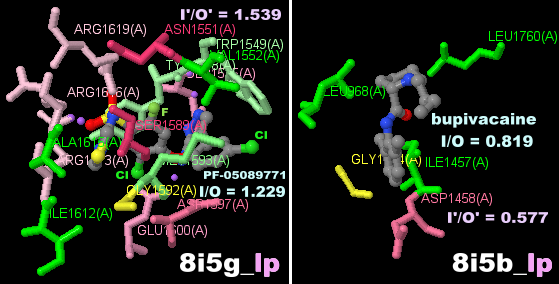
阻害剤PF-05089771が結合した電位依存性ナトリウムチャネルNav1.7 8i5gのChain AとPDBsumデータ(ブピバカイン〈bupivacaine〉結合の8i5bとの比較)
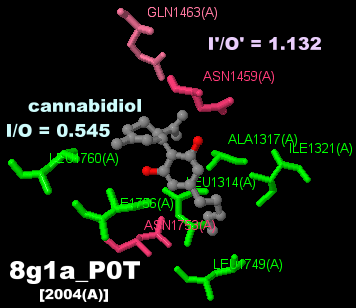
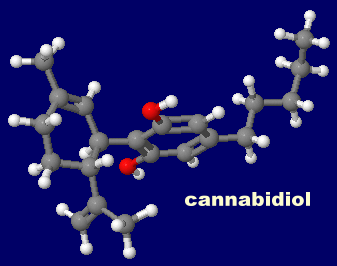
[左・中]阻害剤カンナビジオール(cannabidiol)が結合した電位依存性ナトリウムチャネルNav1.7 8g1aのChain AとPDBsumデータ [右]カンナビジオール(cannabidiol)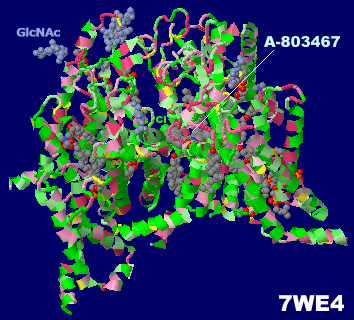
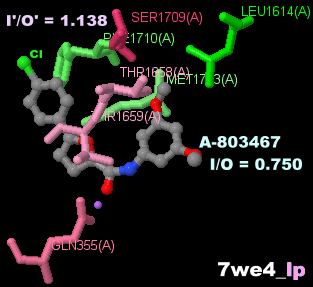
遮断剤A-803467が結合した電位依存性ナトリウムチャネルNav1.8 7we4とPDBsumデータ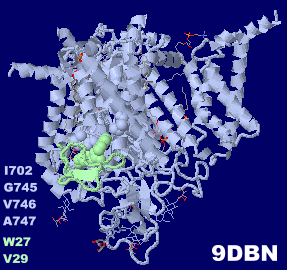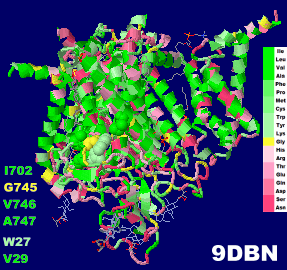
タランチュラ毒Protoxin-Iが結合した電位依存性ナトリウムチャネルNav1.8 9dbn
※球表示したChain AのI702・G745・V746・A747およびChain BのW27・V29は論文参照
Chain Bのアミノ酸配列:I/O値順(特性基 R)着色 [Protoxin-I - Wikipedia]
Glu
Cys
Arg
Tyr
Trp
Leu
Gly
Gly
Cys
Ser
Ala
Gly
Gln
Thr
Cys
Cys
Lys
His
Leu
Val
Cys
Ser
Arg
Arg
His
Gly
Trp
Cys
Val
Trp
Asp
Gly
Thr
Phe
Ser
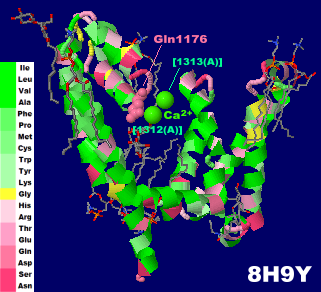
イオンチャネルNavAb(N49K/L176Q変異体) 8h9y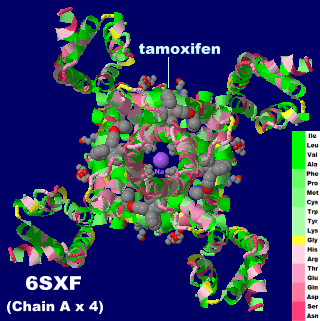
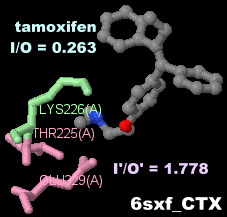
タモキシフェン(tamoxifen)が結合した電位依存性ナトリウムチャネルNavMs 6sxfの4量体(Chain A×4)とPDBsumデータ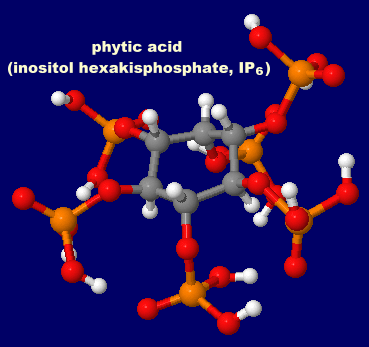
[左]イノシトール6リン酸(IP6)
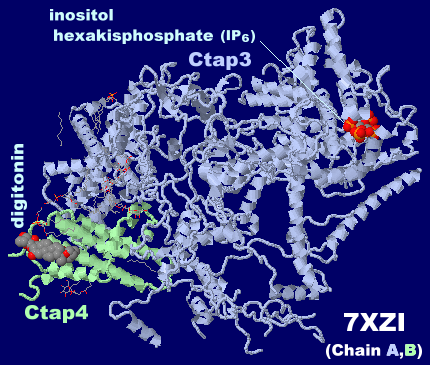
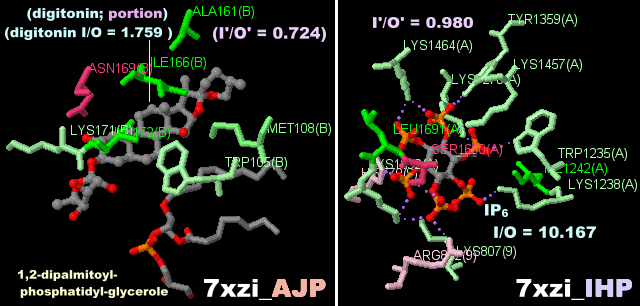
[中・右]参考データ:ジギトニンとイノシトール6リン酸が結合したTOC-TIC超複合体中のCtap3・Ctap4(Ctapは葉緑体トランスロコン関連タンパク質)7xziのChain A・BとPDBsumデータ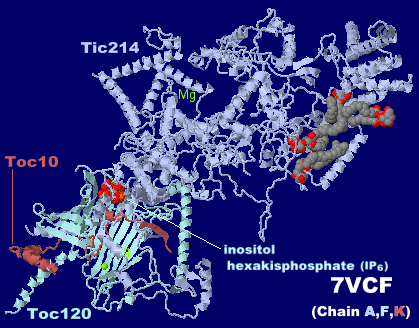
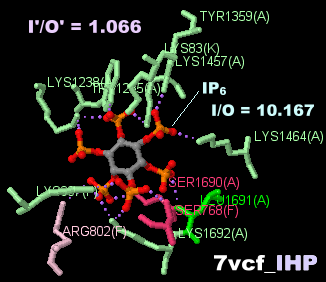
イノシトール6リン酸(IP6)が結合したTOC-TIC超複合体7vcfのChain A・F・K(Tic214・Toc120・Toc10)とPDBsumデータ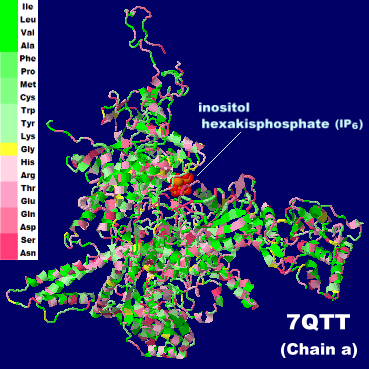
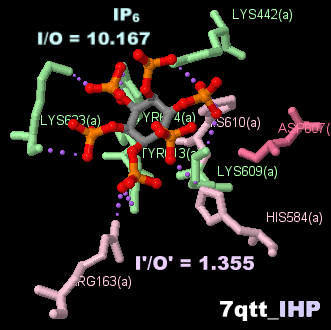
参考データ:イノシトール6リン酸が結合したmRNA前駆体プロセシングスプライシング因子8 7qttのChain aとPDBsumデータ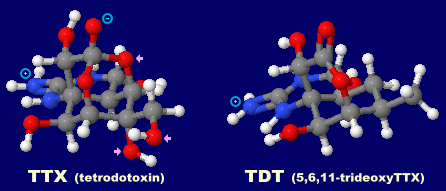
※参考:テトロドトキシン(TTX)とその類縁体例 TDT(5,6,11-trideoxyTTX)
[Chlorophyll f - Wikipedia]
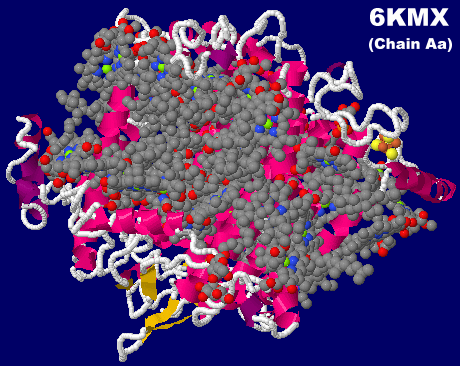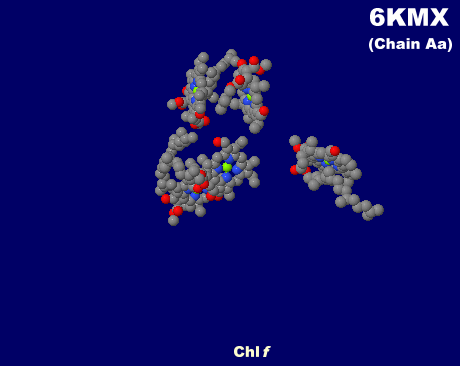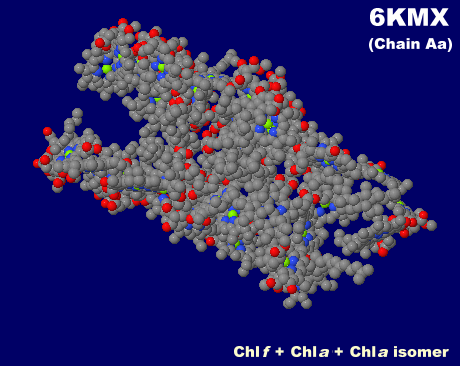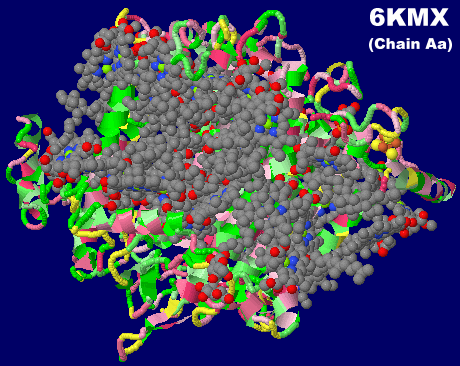
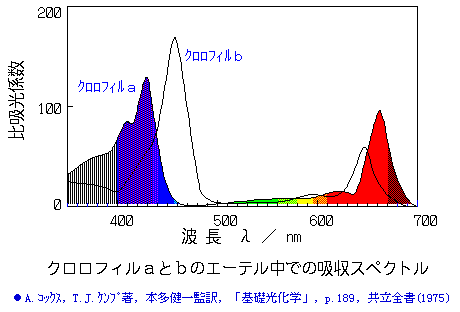
[左]クロロフィルf・a・同アイソマーを含むシアノバクテリアの光化学系I 6kmxのChain Aa [右]参考:クロロフィルa・bの吸収スペクトル例
[Halicin - Wikipedia]
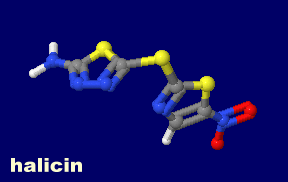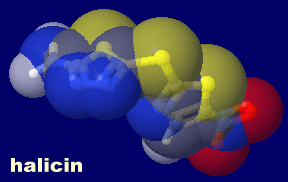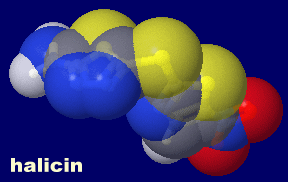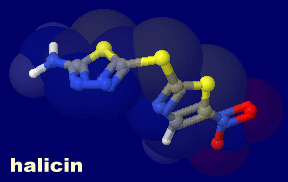
ハリシン(halicin,SU-3327)